ここから本文です
顔面肩甲上腕型筋ジストロフィー(FSHD)原因遺伝子による異常な転写産物を同定 ~FSHDの病態解明に寄与できる可能性を示唆~
2021年06月09日
東海大学工学部生命化学科准教授の三橋弘明、同医学部医学科講師 中川草(両者は本学マイクロ・ナノ研究開発センター兼任)、東京医科歯科大学の三橋里美准教授らの研究グループは、顔面肩甲上腕型筋ジストロフィー(FSHD)の原因遺伝子DUX4による異常な転写産物の同定に成功しました。この研究をまとめた論文が、2021年4月1日(木)に『Human Molecular Genetics』へ掲載されました。
<本件のポイント>
①DUX4-flによって発現誘導される転写産物を最新鋭のロングリードシークエンサーを用いて網羅的に解析した。
②従来のシークエンス技術では解読困難であった反復配列からの転写産物の詳細な解読に成功した。
③DUX4-flが特異的に発現誘導する反復配列はFSHD患者由来のiPS細胞でも発現していた。
■研究の概要
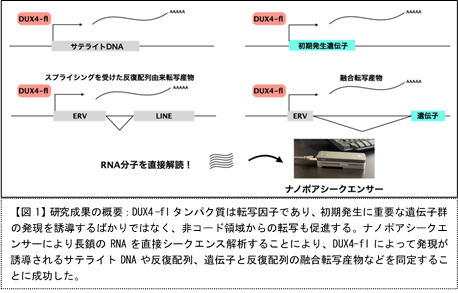
本研究グループは、FSHDの原因となるDUX4-flタンパク質が制御する転写産物*1を最新鋭のロングリードシークエンサー*2を用いて網羅的に解析し、内在性レトロウイルス(ERV)などの遺伝子以外の領域(非コード領域*3)からの異常な転写がDUX4-flによって活性化されることを明らかにしました【図1】。また、FSHD患者由来のiPS細胞においてもこれらの転写産物が発現していたことから、FSHD病態との関連が示唆されます。
この研究は、文部科学省先進ゲノム支援(先進ゲノム解析研究推進プラットフォーム)の支援のもと、東京医科歯科大学、京都大学iPS細胞研究所、産業技術総合研究所との共同研究によって行われました。
■研究の背景
FSHDは常染色体優性(顕性)遺伝形式*4で遺伝する筋疾患で、国の指定難病の1つです。希少疾患である筋ジストロフィーの中では、最も患者数の多い型の1つと推定されています。FSHD患者は主に顔、肩、上腕の筋肉に萎縮と筋力低下を示し、症状は次第に進行していきます。FSHDの遺伝学的な原因は、4番染色体サブテロメア*5にあるD4Z4と呼ばれる反復配列(D4Z4リピート)にあり、患者の約95%ではD4Z4の反復回数が減少しています。D4Z4リピートの末端にはDUX4と呼ばれる遺伝子が存在しており、健常者の筋細胞ではDUX4遺伝子は発現が抑制されていますが、FSHD患者ではD4Z4リピートの短縮が原因でDUX4遺伝子の抑制が弱まっています。そのため、DUX4遺伝子から転写が起こり、選択的スプライシング*6により全長型のDUX4-flと、C末端側が短いDUX4-sの2つの異なる転写因子タンパク質*7が産生されます。特に、FSHD患者の筋肉では全長型のDUX4-flが高頻度に産生されており、このDUX4-flの異所性の発現が筋細胞にさまざまな障害をもたらすと考えられています。DUX4-flはヒトの初期胚に一過性に発現し、多数の初期胚特異的遺伝子の発現を促進することがわかっているため、正常な機能として、初期発生に必要な遺伝子群を調節するマスター転写因子としての役割が考えられています。FSHD患者の骨格筋においてもDUX4-flにより初期胚特異的遺伝子の発現が誘導されていますが、それらがどのようにして筋ジストロフィー症状に関与するのかはまだ解明されていません。
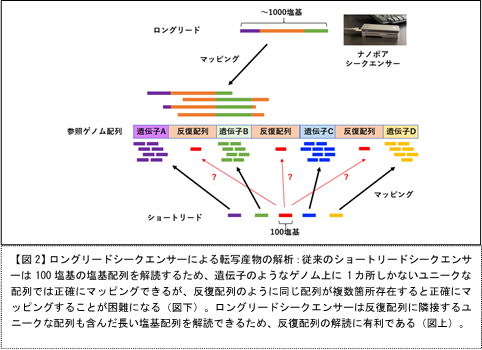
転写因子のDUX4-flはタンパク質を産生する遺伝子のみではなく、遺伝子以外の領域からの転写も促進するとの報告が近年続いています。ヒトのゲノムDNAには遺伝子領域はおよそ1.5%程度しかなく、反復(リピート)配列と呼ばれるものが半分以上を占めていることが知られています。反復配列は互いによく似た塩基配列を持っており、ゲノム上に複数コピーが存在しています。特に内在性レトロウイルス配列(ERV)*8やLINE*9、SINE*10など転移因子に由来する配列や、サテライトDNAと呼ばれる繰り返し配列が、セントロメア*11、テロメアなど非コード領域に多く存在しています。これまでに、DUX4-flがERVやセントロメア付近のサテライトDNAの転写を促進することが報告されていましたが、その正確な座位(locus)や詳細な配列についてはあまりよくわかっていませんでした。その理由は、現在、転写産物の網羅的な解析に一般的に使われているシークエンサーは100塩基程度の短い塩基配列断片を解読するため、ゲノム上に複数、同じ配列がある場合、どの場所から転写が起きているのか、またどの部分がつなぎ合わさっているのかを判別するのが難しいためです。
私たちの研究グループはDUX4-flが制御する反復配列由来の転写産物の全貌を明らかにすることが疾患の機序解明に重要であると考えました。そこで、10,000塩基以上の長い塩基配列を解読することができるロングリードシークエンサーの1つであるナノポアシークエンサー*12を用い、RNA分子を網羅的に直接解読する、ロングリードトランスクリプトーム解析*13を行うことにしました。ナノポアシークエンサーは長い反復配列の解読において、従来のシークエンサーよりも適していると考えられ、転写産物のエキソン構造を解明し、スプライシングアイソフォーム*14を正確に同定することができると期待しました【図2】。
■研究内容
ヒト骨格筋由来の細胞株であるRD細胞にDUX4-fl発現コンストラクトを導入し、RD細胞の中でDUX4-flによって調節されるさまざまな転写産物が産生される状態を作りました。対照実験として、正常な筋細胞でも発現が報告されているDUX4-sを用いて同様の実験を行いました。DUX4を導入した細胞からポリA付加RNAを抽出し、現時点で最も多いデータ量が得られるPromethIONナノポアシークエンサー(Oxford Nanopore Technologies社)を用い、転写産物の網羅的な配列解読を行いました。従来のショートリードシークエンサーでは、RNAから逆転写で合成したDNAを100塩基程度の短い断片として解読するのに対し、本解析では平均800塩基程度のRNAの塩基配列をナノポアシークエンサーで解読することができました。得られたシークエンスデータをLAST(https://gitlab.com/mcfrith/last)を用いて参照ゲノム配列*15へマッピングし、その後last-rna(https://github.com/mcfrith/last-rna)を用いて詳細な配列解析を行いました。
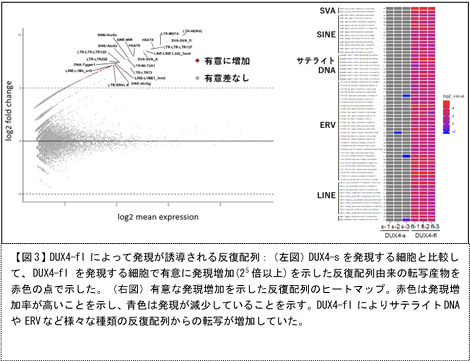
その結果、DUX4-flを導入したRD細胞では、多くの遺伝子の発現が増加していることがわかりました。その多くは初期胚の4細胞期に発現する遺伝子であり、過去の報告と一致していたことから我々のナノポアシークエンシングがDUX4-flによって誘導される転写産物を正しく検出していることが確認できました。さらに、Dfam*16やRepBase*17 などによって定義されている反復配列からの転写を調べたところ、61箇所の座位(locus)からERVなどの反復配列由来の転写産物が明らかに発現誘導されていることがわかりました【図3】。
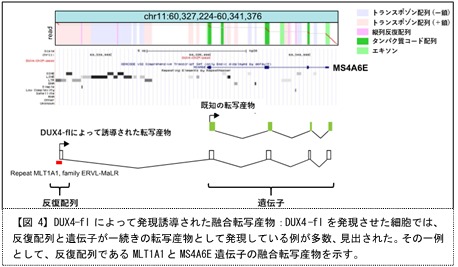
これらはDUX4-flの結合部位(ChIPピーク)とも一致していたことから、DUX4-flによって直接的に発現が調節されたものと考えられます。興味深いことに、これらの反復配列由来の転写産物のほとんどがスプライシングを受けていることがわかりました。また、セントロメア付近のおよそ5箇所の領域から、サテライトDNAが転写されていることも明らかとなりました。予想外にもこれらのサテライトDNA転写産物には、遺伝子から転写されたmRNAと同程度の長さのポリA配列が付加されており、何らかの生理的な意義が示唆されました。さらに興味深いことに、ERV配列と近傍の遺伝子との融合転写産物を247個見いだしました【図4】。
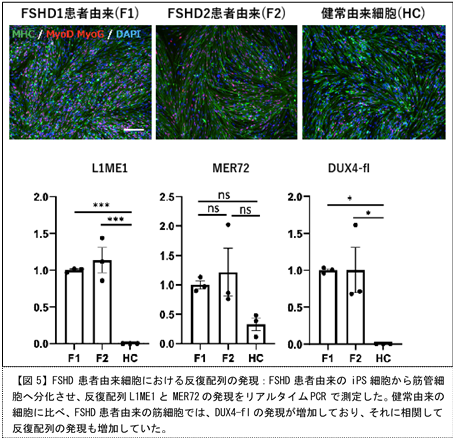
そのうち216個はこれまでに報告のない融合転写産物でした。これらの反復配列からの転写や反復配列を含む融合転写産物はDUX4-sを導入したRD細胞では見られなかったため、DUX4-flによって誘導された異常な転写産物と考えられます。そこでこれらの異常な転写産物がFSHD患者の体で実際に起きているかを確かめるため、FSHD患者由来iPS細胞から分化させた筋細胞を用いて反復配列の検出を行いました。その結果、いくつかの反復配列はFSHD患者由来iPS細胞においても発現が確認されました【図5】。
■本研究の意義と今後の展望
DUX4-flがどのような機序で筋肉に障害をもたらすのかは現在のところ解明されていませんが、DUX4-flによって制御されている転写産物を明らかにすることが謎を解く1つの鍵になると考えられます。今回、反復配列を含む多くの転写産物の座位や発現量、詳細な塩基配列を明らかにすることができたため、今後、これらの転写産物の機能を追求することでFSHD病態の解明に寄与できる可能性があると期待しています。また、これまでにゲノムDNAの反復配列の解読にナノポアシークエンサーを活用した報告はありましたが、反復配列由来の転写産物を網羅的に解析した報告はなく、世界に先駆けた成果となりました。本研究で用いた手法は、今後、FSHDの研究だけではなく、初期発生やトランスポゾンの発現が関与する様々な生命現象や疾患への応用が期待できます
【掲載論文】
雑誌名 : 『Human Molecular Genetics』 (2021年4月1日掲載)
タイトル : Nanopore direct RNA sequencing detects DUX4-activated repeats and isoforms in human muscle cells.
URL : https://academic.oup.com/hmg/article-abstract/30/7/552/6160822
DOI : 10.1093/hmg/ddab063
【筆者】
三橋里美1,2、中川草3,4、本田 充(佐々木充)5、櫻井英俊5、フリス マーティン6,7,8、三橋弘明3,9*
1. 東京医科歯科大学難治疾患研究所
2. 横浜市立大学医学部医学科
3. 東海大学マイクロ・ナノ研究開発センター
4. 東海大学医学部医学科
5. 京都大学iPS細胞研究所
6. 国立研究開発法人産業技術総合研究所人工知能研究センター
7. 東京大学大学院新領域創成科学研究科
8. 産総研・早大生体システムビッグデータ解析オープンイノベーションラボラトリ
9. 東海大学工学部生命化学科
*責任著者
用語解説
*1転写産物:ゲノムDNAを鋳型として合成されるRNAの総称。
*2 ロングリードシークエンサー:1万塩基以上の核酸の塩基配列を一つづきに解読することができる装置。本研究ではOxford Nanopore Technologies社の装置PromethIONを用いた。
*3 非コード領域:DNAはグアニン(G)、シトシン(C)、アデニン(A)、チミン (T)の4種類の塩基の配列で情報を担う遺伝物質であり、タンパク質を産生するための情報を持つ配列を遺伝子と呼ぶ。遺伝子からは転写によりmRNAが合成され、mRNAが翻訳されてタンパク質が合成される。それに対し、タンパク質を産生するための情報を持っていないゲノムDNA領域を総称して非コード領域と呼ぶ。非コード領域の大部分を反復配列が占めている。一部の非コード領域では転写が起こり、RNAが合成されることがわかっているが、RNAは翻訳されずタンパク質は産生されないと考えられている。非コード領域から転写されるRNAの生理的な意義はまだ不明な点が多い。
*4 常染色体優性(顕性)遺伝形式:常染色体は性別に関係なく細胞に2本1組で存在する染色体であり、人間では1番染色体から22番染色体までの22組(44本)を指す。2本1組であるのは、両親から1本ずつ受け継ぐためである(例を挙げると、父親由来の1番染色体と母親由来の1番染色体があるため、1番染色体を2本1組で持つことになる)。そのため、人間は常染色体上にある遺伝子を2つ1組で持つ。1組の遺伝子のうち、片方の遺伝子に変化(変異)が生じただけで病気を発症する場合、優性遺伝形式の疾患と呼ぶ。顕性遺伝形式とも言う。これに対して、1組の遺伝子の両方に変異が生じた時にはじめて病気を発症する場合は、劣性(潜性)遺伝形式と言う。
*5 サブテロメア:染色体の末端にはテロメアと呼ばれる特別な繰り返し配列が存在する。サブテロメアはテロメアに隣接する領域。
*6 選択的スプライシング:遺伝子から転写によって合成された一次転写産物には、イントロンと呼ばれる遺伝情報を含まない配列を含む場合が多い。このイントロンを除く過程をスプライシングと呼ぶ。スプライシングを行う部位の変化により、1つの遺伝子から複数の成熟mRNAが生成されることがあり、これを選択的スプライシングと呼ぶ。
*7 転写因子タンパク質:DNAの特定の塩基配列を認識して結合し、DNAの遺伝情報をRNAに転写する過程を促進、あるいは抑制する機能を持つタンパク質の一群。いつ、どの細胞でどのくらいRNAを合成するかという遺伝子発現の調節を担っている。
*8 内在性レトロウイルス配列(ERV):ヒトを含めた様々な生物のゲノムDNAの中にあるレトロウイルスに類似した配列。ヒトゲノム全体のおよそ8%がERVである。生殖細胞系列に感染したレトロウイルスの配列がもととなっていると考えられている。逆転写酵素と呼ばれるRNAからDNAに変換できる酵素を産生し、ゲノム内を増殖しながら移動するレトロトランスポゾンの一種である。
*9 LINE:ERVと同様のレトロトランスポゾンの一種であり、逆転写酵素を産生してゲノム内を増殖しながら移動するが、レトロウイルスとは構造が異なる。ヒトゲノム全体のおよそ20%がLINEである。
*10 SINE:逆転写酵素やタンパク質は産生しないが、LINEの逆転写酵素を利用してゲノム内を増殖しながら移動するレトロトランスポゾンの一種。ヒトゲノムの13%前後がSINEに対応する。
*11 セントロメア:染色体の長腕と短腕が交差する部位。特殊な繰り返し配列が存在する。
*12 ナノポアシークエンサー:ナノポアというタンパク質の穴が人工膜に埋め込まれており、ナノポアを核酸分子が通り抜ける時に起きる電流の変化によって、塩基配列を解読するタイプのシークエンサー。一般的な転写産物解析ではRNAから相補的なDNAを合成し塩基配列を解読するが、ナノポアシークエンサーでは直接RNAの塩基配列の解読が可能である。
*13 ロングリードトランスクリプトーム解析:ロングリードシークエンサーを用いて、細胞の全転写産物を解析する手法。
*14 スプライシングアイソフォーム:選択的スプライシングによって産生された異なる種類のRNA
*15 参照ゲノム配列:国際プロジェクトによって解読された人間のゲノムDNAの全塩基配列で、解析の際に基準となる配列。ゲノムDNAの塩基配列は個人ごとに少しずつ異なるため、各々の研究で解読した塩基配列を参照ゲノム配列に照らし合わせることで解析を行う。
*16 Dfam:ゲノムに存在する反復配列を収集したデータベース、データは公開されている。
*17 RepBase:ゲノムに存在する反復配列を収集した世界最大のデータベース、データの利用には登録が必要(有料)である。
■本件に関するお問い合わせ
東海大学工学部生命化学科 担当:三橋弘明
TEL.0463-58-1211(代表)
E-mail:hmitsuhashi@tsc.u-tokai.ac.jp
<本件のポイント>
①DUX4-flによって発現誘導される転写産物を最新鋭のロングリードシークエンサーを用いて網羅的に解析した。
②従来のシークエンス技術では解読困難であった反復配列からの転写産物の詳細な解読に成功した。
③DUX4-flが特異的に発現誘導する反復配列はFSHD患者由来のiPS細胞でも発現していた。
■研究の概要
本研究グループは、FSHDの原因となるDUX4-flタンパク質が制御する転写産物*1を最新鋭のロングリードシークエンサー*2を用いて網羅的に解析し、内在性レトロウイルス(ERV)などの遺伝子以外の領域(非コード領域*3)からの異常な転写がDUX4-flによって活性化されることを明らかにしました【図1】。また、FSHD患者由来のiPS細胞においてもこれらの転写産物が発現していたことから、FSHD病態との関連が示唆されます。
この研究は、文部科学省先進ゲノム支援(先進ゲノム解析研究推進プラットフォーム)の支援のもと、東京医科歯科大学、京都大学iPS細胞研究所、産業技術総合研究所との共同研究によって行われました。
■研究の背景
FSHDは常染色体優性(顕性)遺伝形式*4で遺伝する筋疾患で、国の指定難病の1つです。希少疾患である筋ジストロフィーの中では、最も患者数の多い型の1つと推定されています。FSHD患者は主に顔、肩、上腕の筋肉に萎縮と筋力低下を示し、症状は次第に進行していきます。FSHDの遺伝学的な原因は、4番染色体サブテロメア*5にあるD4Z4と呼ばれる反復配列(D4Z4リピート)にあり、患者の約95%ではD4Z4の反復回数が減少しています。D4Z4リピートの末端にはDUX4と呼ばれる遺伝子が存在しており、健常者の筋細胞ではDUX4遺伝子は発現が抑制されていますが、FSHD患者ではD4Z4リピートの短縮が原因でDUX4遺伝子の抑制が弱まっています。そのため、DUX4遺伝子から転写が起こり、選択的スプライシング*6により全長型のDUX4-flと、C末端側が短いDUX4-sの2つの異なる転写因子タンパク質*7が産生されます。特に、FSHD患者の筋肉では全長型のDUX4-flが高頻度に産生されており、このDUX4-flの異所性の発現が筋細胞にさまざまな障害をもたらすと考えられています。DUX4-flはヒトの初期胚に一過性に発現し、多数の初期胚特異的遺伝子の発現を促進することがわかっているため、正常な機能として、初期発生に必要な遺伝子群を調節するマスター転写因子としての役割が考えられています。FSHD患者の骨格筋においてもDUX4-flにより初期胚特異的遺伝子の発現が誘導されていますが、それらがどのようにして筋ジストロフィー症状に関与するのかはまだ解明されていません。
転写因子のDUX4-flはタンパク質を産生する遺伝子のみではなく、遺伝子以外の領域からの転写も促進するとの報告が近年続いています。ヒトのゲノムDNAには遺伝子領域はおよそ1.5%程度しかなく、反復(リピート)配列と呼ばれるものが半分以上を占めていることが知られています。反復配列は互いによく似た塩基配列を持っており、ゲノム上に複数コピーが存在しています。特に内在性レトロウイルス配列(ERV)*8やLINE*9、SINE*10など転移因子に由来する配列や、サテライトDNAと呼ばれる繰り返し配列が、セントロメア*11、テロメアなど非コード領域に多く存在しています。これまでに、DUX4-flがERVやセントロメア付近のサテライトDNAの転写を促進することが報告されていましたが、その正確な座位(locus)や詳細な配列についてはあまりよくわかっていませんでした。その理由は、現在、転写産物の網羅的な解析に一般的に使われているシークエンサーは100塩基程度の短い塩基配列断片を解読するため、ゲノム上に複数、同じ配列がある場合、どの場所から転写が起きているのか、またどの部分がつなぎ合わさっているのかを判別するのが難しいためです。
私たちの研究グループはDUX4-flが制御する反復配列由来の転写産物の全貌を明らかにすることが疾患の機序解明に重要であると考えました。そこで、10,000塩基以上の長い塩基配列を解読することができるロングリードシークエンサーの1つであるナノポアシークエンサー*12を用い、RNA分子を網羅的に直接解読する、ロングリードトランスクリプトーム解析*13を行うことにしました。ナノポアシークエンサーは長い反復配列の解読において、従来のシークエンサーよりも適していると考えられ、転写産物のエキソン構造を解明し、スプライシングアイソフォーム*14を正確に同定することができると期待しました【図2】。
■研究内容
ヒト骨格筋由来の細胞株であるRD細胞にDUX4-fl発現コンストラクトを導入し、RD細胞の中でDUX4-flによって調節されるさまざまな転写産物が産生される状態を作りました。対照実験として、正常な筋細胞でも発現が報告されているDUX4-sを用いて同様の実験を行いました。DUX4を導入した細胞からポリA付加RNAを抽出し、現時点で最も多いデータ量が得られるPromethIONナノポアシークエンサー(Oxford Nanopore Technologies社)を用い、転写産物の網羅的な配列解読を行いました。従来のショートリードシークエンサーでは、RNAから逆転写で合成したDNAを100塩基程度の短い断片として解読するのに対し、本解析では平均800塩基程度のRNAの塩基配列をナノポアシークエンサーで解読することができました。得られたシークエンスデータをLAST(https://gitlab.com/mcfrith/last)を用いて参照ゲノム配列*15へマッピングし、その後last-rna(https://github.com/mcfrith/last-rna)を用いて詳細な配列解析を行いました。
その結果、DUX4-flを導入したRD細胞では、多くの遺伝子の発現が増加していることがわかりました。その多くは初期胚の4細胞期に発現する遺伝子であり、過去の報告と一致していたことから我々のナノポアシークエンシングがDUX4-flによって誘導される転写産物を正しく検出していることが確認できました。さらに、Dfam*16やRepBase*17 などによって定義されている反復配列からの転写を調べたところ、61箇所の座位(locus)からERVなどの反復配列由来の転写産物が明らかに発現誘導されていることがわかりました【図3】。
これらはDUX4-flの結合部位(ChIPピーク)とも一致していたことから、DUX4-flによって直接的に発現が調節されたものと考えられます。興味深いことに、これらの反復配列由来の転写産物のほとんどがスプライシングを受けていることがわかりました。また、セントロメア付近のおよそ5箇所の領域から、サテライトDNAが転写されていることも明らかとなりました。予想外にもこれらのサテライトDNA転写産物には、遺伝子から転写されたmRNAと同程度の長さのポリA配列が付加されており、何らかの生理的な意義が示唆されました。さらに興味深いことに、ERV配列と近傍の遺伝子との融合転写産物を247個見いだしました【図4】。
そのうち216個はこれまでに報告のない融合転写産物でした。これらの反復配列からの転写や反復配列を含む融合転写産物はDUX4-sを導入したRD細胞では見られなかったため、DUX4-flによって誘導された異常な転写産物と考えられます。そこでこれらの異常な転写産物がFSHD患者の体で実際に起きているかを確かめるため、FSHD患者由来iPS細胞から分化させた筋細胞を用いて反復配列の検出を行いました。その結果、いくつかの反復配列はFSHD患者由来iPS細胞においても発現が確認されました【図5】。
■本研究の意義と今後の展望
DUX4-flがどのような機序で筋肉に障害をもたらすのかは現在のところ解明されていませんが、DUX4-flによって制御されている転写産物を明らかにすることが謎を解く1つの鍵になると考えられます。今回、反復配列を含む多くの転写産物の座位や発現量、詳細な塩基配列を明らかにすることができたため、今後、これらの転写産物の機能を追求することでFSHD病態の解明に寄与できる可能性があると期待しています。また、これまでにゲノムDNAの反復配列の解読にナノポアシークエンサーを活用した報告はありましたが、反復配列由来の転写産物を網羅的に解析した報告はなく、世界に先駆けた成果となりました。本研究で用いた手法は、今後、FSHDの研究だけではなく、初期発生やトランスポゾンの発現が関与する様々な生命現象や疾患への応用が期待できます
【掲載論文】
雑誌名 : 『Human Molecular Genetics』 (2021年4月1日掲載)
タイトル : Nanopore direct RNA sequencing detects DUX4-activated repeats and isoforms in human muscle cells.
URL : https://academic.oup.com/hmg/article-abstract/30/7/552/6160822
DOI : 10.1093/hmg/ddab063
【筆者】
三橋里美1,2、中川草3,4、本田 充(佐々木充)5、櫻井英俊5、フリス マーティン6,7,8、三橋弘明3,9*
1. 東京医科歯科大学難治疾患研究所
2. 横浜市立大学医学部医学科
3. 東海大学マイクロ・ナノ研究開発センター
4. 東海大学医学部医学科
5. 京都大学iPS細胞研究所
6. 国立研究開発法人産業技術総合研究所人工知能研究センター
7. 東京大学大学院新領域創成科学研究科
8. 産総研・早大生体システムビッグデータ解析オープンイノベーションラボラトリ
9. 東海大学工学部生命化学科
*責任著者
用語解説
*1転写産物:ゲノムDNAを鋳型として合成されるRNAの総称。
*2 ロングリードシークエンサー:1万塩基以上の核酸の塩基配列を一つづきに解読することができる装置。本研究ではOxford Nanopore Technologies社の装置PromethIONを用いた。
*3 非コード領域:DNAはグアニン(G)、シトシン(C)、アデニン(A)、チミン (T)の4種類の塩基の配列で情報を担う遺伝物質であり、タンパク質を産生するための情報を持つ配列を遺伝子と呼ぶ。遺伝子からは転写によりmRNAが合成され、mRNAが翻訳されてタンパク質が合成される。それに対し、タンパク質を産生するための情報を持っていないゲノムDNA領域を総称して非コード領域と呼ぶ。非コード領域の大部分を反復配列が占めている。一部の非コード領域では転写が起こり、RNAが合成されることがわかっているが、RNAは翻訳されずタンパク質は産生されないと考えられている。非コード領域から転写されるRNAの生理的な意義はまだ不明な点が多い。
*4 常染色体優性(顕性)遺伝形式:常染色体は性別に関係なく細胞に2本1組で存在する染色体であり、人間では1番染色体から22番染色体までの22組(44本)を指す。2本1組であるのは、両親から1本ずつ受け継ぐためである(例を挙げると、父親由来の1番染色体と母親由来の1番染色体があるため、1番染色体を2本1組で持つことになる)。そのため、人間は常染色体上にある遺伝子を2つ1組で持つ。1組の遺伝子のうち、片方の遺伝子に変化(変異)が生じただけで病気を発症する場合、優性遺伝形式の疾患と呼ぶ。顕性遺伝形式とも言う。これに対して、1組の遺伝子の両方に変異が生じた時にはじめて病気を発症する場合は、劣性(潜性)遺伝形式と言う。
*5 サブテロメア:染色体の末端にはテロメアと呼ばれる特別な繰り返し配列が存在する。サブテロメアはテロメアに隣接する領域。
*6 選択的スプライシング:遺伝子から転写によって合成された一次転写産物には、イントロンと呼ばれる遺伝情報を含まない配列を含む場合が多い。このイントロンを除く過程をスプライシングと呼ぶ。スプライシングを行う部位の変化により、1つの遺伝子から複数の成熟mRNAが生成されることがあり、これを選択的スプライシングと呼ぶ。
*7 転写因子タンパク質:DNAの特定の塩基配列を認識して結合し、DNAの遺伝情報をRNAに転写する過程を促進、あるいは抑制する機能を持つタンパク質の一群。いつ、どの細胞でどのくらいRNAを合成するかという遺伝子発現の調節を担っている。
*8 内在性レトロウイルス配列(ERV):ヒトを含めた様々な生物のゲノムDNAの中にあるレトロウイルスに類似した配列。ヒトゲノム全体のおよそ8%がERVである。生殖細胞系列に感染したレトロウイルスの配列がもととなっていると考えられている。逆転写酵素と呼ばれるRNAからDNAに変換できる酵素を産生し、ゲノム内を増殖しながら移動するレトロトランスポゾンの一種である。
*9 LINE:ERVと同様のレトロトランスポゾンの一種であり、逆転写酵素を産生してゲノム内を増殖しながら移動するが、レトロウイルスとは構造が異なる。ヒトゲノム全体のおよそ20%がLINEである。
*10 SINE:逆転写酵素やタンパク質は産生しないが、LINEの逆転写酵素を利用してゲノム内を増殖しながら移動するレトロトランスポゾンの一種。ヒトゲノムの13%前後がSINEに対応する。
*11 セントロメア:染色体の長腕と短腕が交差する部位。特殊な繰り返し配列が存在する。
*12 ナノポアシークエンサー:ナノポアというタンパク質の穴が人工膜に埋め込まれており、ナノポアを核酸分子が通り抜ける時に起きる電流の変化によって、塩基配列を解読するタイプのシークエンサー。一般的な転写産物解析ではRNAから相補的なDNAを合成し塩基配列を解読するが、ナノポアシークエンサーでは直接RNAの塩基配列の解読が可能である。
*13 ロングリードトランスクリプトーム解析:ロングリードシークエンサーを用いて、細胞の全転写産物を解析する手法。
*14 スプライシングアイソフォーム:選択的スプライシングによって産生された異なる種類のRNA
*15 参照ゲノム配列:国際プロジェクトによって解読された人間のゲノムDNAの全塩基配列で、解析の際に基準となる配列。ゲノムDNAの塩基配列は個人ごとに少しずつ異なるため、各々の研究で解読した塩基配列を参照ゲノム配列に照らし合わせることで解析を行う。
*16 Dfam:ゲノムに存在する反復配列を収集したデータベース、データは公開されている。
*17 RepBase:ゲノムに存在する反復配列を収集した世界最大のデータベース、データの利用には登録が必要(有料)である。
■本件に関するお問い合わせ
東海大学工学部生命化学科 担当:三橋弘明
TEL.0463-58-1211(代表)
E-mail:hmitsuhashi@tsc.u-tokai.ac.jp