ここから本文です
新型コロナウイルスnsp14遺伝子の変異はゲノム多様性の向上につながる ~ゲノムデータ解析とウイルス実験によりコロナウイルスの進化の一端を解明~
2023年03月24日
東海大学[伊勢原校舎]医学部医学科分子生命科学の中川草准教授では、大阪大学微生物病研究所分子ウイルス分野の渡辺登喜子教授らとの共同研究で、新型コロナウイルスのnsp14*1遺伝子の変異はゲノム多様性を向上させる可能性があることをデータ解析とウイルス実験で解明しました。この研究成果が、2023年2月15日(水)、「iScience」(オンライン版)に掲載されました。
|
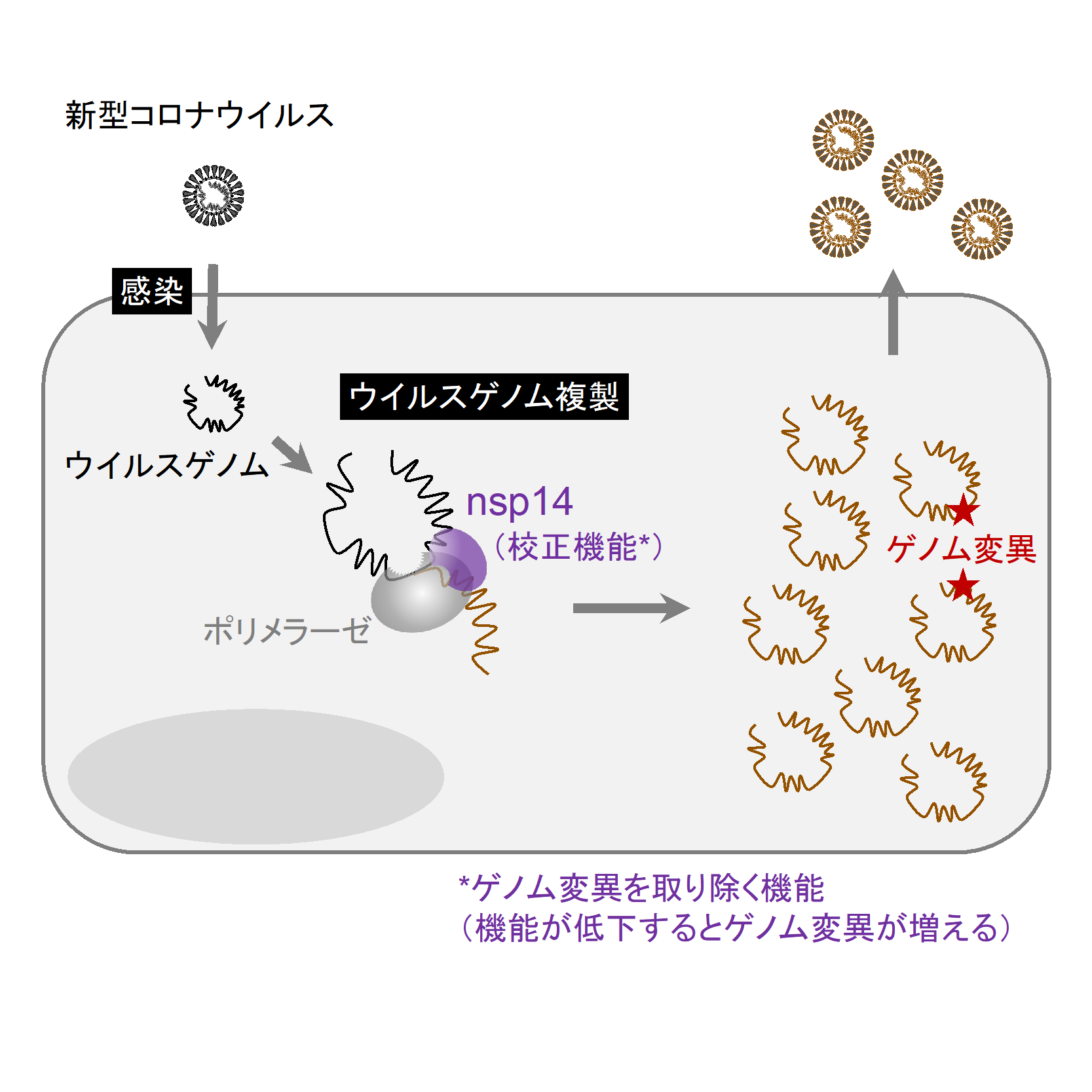
研究のポイント ・コロナウイルスはゲノム複製時のエラーを校正するウイルスタンパク質 (nsp14:図1)を持ち、さまざまなコロナウイルスウイルス(コウモリ由来のコロナウイルスなど)で高く保存されている領域が多いことが判明。 ・新型コロナウイルスのゲノムデータ解析により、nsp14-P203Lを持つウイルス株は、ゲノム変異しやすい傾向を持つことを発見。 ・動物実験により、ハムスターの肺で増えた変異型nsp14-P203Lウイルスには、多様なゲノム変異が見られることを解明。(図2)。 ・nsp14の機能維持は、新型コロナウイルスにおいて重要である可能性が示唆された。 |
■研究の概要
ウイルスのゲノムに生じる突然変異は、ウイルスの進化と密接に関係しています。一般的にRNAウイルスは、ゲノム複製時にゲノム変異を起こしやすいことが知られています。これはゲノム、つまり遺伝子を変異させることで、常に変化する環境に適応するためのウイルスの生存戦略ともいえます。ゲノムの変異によって、感染性や増殖能が向上することもあれば、逆に増殖できなくなってしまうような変異もあります。特に、RNAウイルスの一つであるコロナウイルスは、インフルエンザウイルスなど他のRNAウイルスに比べて大きなゲノムを持つことから、そのゲノム変異がウイルス生存に重篤な影響をもたらしかねないことから、ゲノム複製時のエラー校正機能を担うウイルスタンパク質(nsp14)を持っています。そのため、インフルエンザ等の他のRNAウイルスと比較して、ゲノム変異が起こりにくいと考えられています。しかし、2019年に出現した新型コロナウイルスは多様な変異ウイルスのゲノム配列が解読されているものもあり、そのなかにはnsp14の校正機能が低下した新型コロナウイルスが存在するのではないかと考え、そのようなnsp14変異株を探索しました。
nsp14の機能に重要なアミノ酸サイトを調べるため、コロナウイルス科に属する62種類のコロナウイルスのnsp14のアミノ酸配列をアライメントし、分子進化解析を行いました。その結果、コロナウイルスのnsp14は進化的によく保存されており、強い負の自然選択*2を受けていることが明らかとなりました。次に、新型コロナウイルスの大規模ゲノムデータ解析を実施したところ、懸念すべき変異株(アルファ株、デルタ株、オミクロン株など)の出現前では、92%以上のウイルス株で同じアミノ酸配列のnsp14(野生型nsp14)を持ち、一方で、コウモリ由来の多様なコロナウイルスにおいても、検出されていないnsp14のアミノ酸置換を持つものもありました。このようなアミノ酸置換を持つ変異株に着目し、そのゲノム進化速度を解析したところ、nsp14-P203Lを持つウイルス株はゲノム進化速度が大きい傾向があることを発見しました。
そこで、nsp14-P203Lがウイルス性状に及ぼす影響を調べるために、リバースジェネティクスを用いて、nsp14-P203Lを持つウイルス(変異型nsp14ウイルス)または野生型nsp14を持つウイルス(野生型nsp14ウイルス)を作出しました。作出したウイルスをハムスターに感染させ、肺で増殖したウイルスのゲノム変異を解析したところ、変異型nsp14ウイルスは野生型nsp14ウイルスよりも多様なゲノム変異が見られることが分かりました。
本研究により、変異型nsp14(例えばnsp14-P203Lなど)が新型コロナウイルスのゲノム多様性を増加させることが分かりました。同時に、このような校正機能が低下した変異型nsp14(例えばnsp14-P203Lなど)を持つウイルスの出現は稀であることも分かりました。実際に、コウモリ由来の多様なコロナウイルスでもnsp14は高く保存されているため、nsp14のゲノム複製エラーを修正する機能は新型コロナウイルスでも重要であると考えています。また、現時点までにnsp14の変異が要因となって公衆衛生上の影響が懸念される変異株が発生しているということは確認されていません。
本研究により、コロナウイルスのnsp14が、コロナウイルス特有の大きなゲノムの維持に重要な役割を持つと同時に、nsp14の機能を維持することは、コロナウイルスの適応と進化に重要であることが明らかになりました。これは、ゲノムデータ解析とウイルス実験を組み合わせることで、ウイルスの進化の一端を紐解いた研究成果ともいえます。この研究手法は、ウイルスの遺伝子変異によるウイルスの性状変化を明らかにする上で非常に有効な手法となることが期待されます。
■本研究への支援
日本学術振興会 新学術研究領域「ネオウイルス学」(16H06429、16K21723、JP16K21723、JP16H06432、16H06434、19H04843)、基盤研究B(JP22H02521、JP21H02736)、研究活動スタート支援(22K15469)、日本学術振興会特別研究員奨励費(21J01036)、科学技術振興機構(JST) 戦略的創造研究推進事業 CREST(JPMJCR20H6)、ムーンショット型研究開発事業(JPMJMS2025)、日本医療研究開発機構(AMED) 新興・再興感染症に対する革新的医薬品等開発推進研究事業(JP20fk0108281、JP19fk0108113、JP20fk0108401、JP21fk0108493、19fk0108171)、医療研究開発革新基盤創成事業(JP20pc0101047)、ワクチン開発のための世界トップレベル研究開発拠点の形成事業(JP223fa627002)、AMED CREST (JP22gm1610010)、東京生化学研究会、武田科学振興財団、リカケンホールディング株式会社などの支援を受けて行われました。
■掲載論文
|
掲載誌 |
: |
iScience (オンライン版) |
|
タイトル |
: |
Genomic diversity of SARS-CoV-2 can be accelerated by mutations in the nsp14 gene |
|
掲載日 |
: |
2023年2月15日(水) |
|
著者 |
: |
高田光輔1,2、上田真保子3、七戸新太郎1、木田裕里恵1、小野慎子4,5、 松浦善治4,5、渡辺登喜子1,4,5*、中川草2,6*(*責任著者) |
|
所属情報 |
|
1.大阪大学微生物病研究所 2.東海大学医学部医学科分子生命科学 3.東京医科歯科大学難治疾患研究所 4.大阪大学感染症総合教育研究拠点 5.大阪大学ワクチン開発拠点先端モダリティ・DDS研究センター 6.国立遺伝学研究所生命情報・DDBJセンター |
|
DOI |
: |
10.1016/j.isci.2023.106210 |
|
URL |
: |
https://www.cell.com/iscience/fulltext/S2589-0042(23)00287-0 |
■用語解説
*1)nsp14
コロナウイルスの非構造蛋白質(non-structural protein)の一つで、コロナウイルスのRNA複製1111複合体の構成要素である。RNA複製時に誤った塩基をもつRNAが付加された場合、それを取り除く校正機能がある。
*2)負の自然選択
個体が次世代に貢献する度合いにばらつきがあり、それが遺伝的に決められているとき、自然選択は生じる。集団に生じる突然変異がその遺伝子をもつ個体の生存力や増殖は損なう場合、そのような有害遺伝子は集団から除去されるという現象を負の選択(淘汰)、もしくは浄化淘汰と呼ぶ。
|
<研究に関するお問い合わせ> 東海大学医学部医学科 分子生命科学 准教授 中川 草(なかがわ そう) TEL.0463-93-1121 内線 2661 E-mail:so@tokai.ac.jp
大阪大学微生物病研究所 分子ウイルス分野 教授 渡辺 登喜子(わたなべ ときこ) TEL.06-6879-8302、FAX:06-6879-8303 E-mail:tokikow@biken.osaka-u.ac.jp
<本件に関するお問い合わせ> 東海大学 ビーワンオフィス 担当:喜友名(、林 TEL.0463-63-4670(直通) E-mail:pr@tsc.u-tokai.ac.jp
大阪大学 微生物病研究所 企画広報推進室 TEL.06-6879-8357、FAX:06-6879-8360 E-mail:biken-pr@biken.osaka-u.ac.jp |