ここから本文です
東海大学医学部 准教授 大塚正人を中心とする研究グループ ノックインマウス、コンディショナルノックアウトマウス作製の高効率化を実現した手法を開発-現在主流となっている「CRISPRゲノム編集法」の課題を解消-
2017年05月18日
東海大学医学部医学科基礎医学系 准教授 大塚 正人をはじめ、米国ネブラスカ大学、東京医科歯科大学の共同研究グループは、この度長鎖一本鎖DNAとCRISPRリボ核タンパク質をツールとして用いることによって、遺伝子改変マウスの作製効率を従来の「CRISPRゲノム編集法*1」に比べ、10倍以上向上させることが可能な手法の開発に成功しました。
なお、本研究成果は5月17日(水)午前1時(イギリス時間)のオンラインジャーナル「Genome Biology」(DOI: 10.1186/s13059-017-1220-4)に掲載されました。
■研究の背景について
遺伝子改変マウスは、個体レベルでの遺伝子機能解析実験や疾患モデル動物として使用される極めて重要なツールの一つです。中でも最も使用頻度の高いマウスは、レポーター遺伝子や組み換え酵素遺伝子などを挿入した「ノックインマウス」、組織・時期特異的に遺伝子を破壊した「コンディショナルノックアウトマウス」であり、作製される全てのマウスの大半をこの二つのマウスが占めています。
これらのマウスを作製するには、標的とする遺伝子座位に対して目的のDNA配列を挿入(ノックイン)する必要があり、最近までES細胞を介した「遺伝子ターゲティング法」によるノックインマウスの作製が主流となっていましたが、この手法では時間とコスト、そして労力を要することが大きな課題となっていました。
こうした課題を受け、近年開発された「CRISPRゲノム編集法」は、受精卵に溶液を注入するだけで、ES細胞を介することなく簡便に目的とする遺伝子改変マウスを作製することが可能な画期的な手法です。しかし、この手法では単純なノックアウトマウスは高効率で作製できる一方で、ノックインマウスの作製効率は優れた技術を有する施設でも高々10%程度、コンディショナルノックアウトマウスでは数%程度と低く、その効率向上が課題となっていました。
「CRISPRゲノム編集法」では、遺伝子をノックインする際に用いる導入DNAに一本鎖DNA*2を用いると効率が高くなりますが、入手可能な一本鎖DNAの長さは200塩基までであり、それ以上の長さの配列を挿入するためには二本鎖DNA*3を用いる必要がありました。しかし、二本鎖DNAを用いた場合のノックイン効率は低く、また比較的長い相同領域*4を付加する必要があることから、ベクターの作製にも労力を要するという課題があります。
ノックイン効率の向上という課題の解消を目指して、いくつかの方法が世界中の様々な研究者によって開発されてきましたが、実用化に至る水準に達しておらず、この課題は遺伝子改変マウス作製に関わる研究分野全体の大きな問題となっており、新たな手法の確立が望まれていました。
■今回の研究成果について
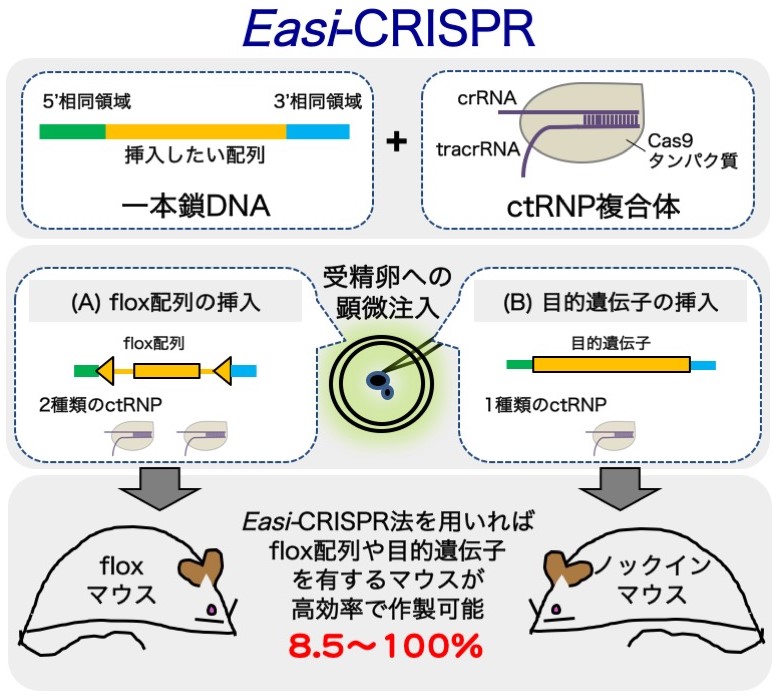
今回、本研究グループでは、長鎖一本鎖DNAとCRISPRリボ核タンパク質を用いることで、受精卵への顕微注入を介したノックイン効率の大幅な向上に成功しました。長鎖一本鎖DNAは、挿入したいDNA配列を有するドナーDNAであり、目的DNA配列の両端に短い相同領域(50~100塩基程度)が付加されています。
本研究グループでは、長い一本鎖DNAを合成する方法を開発し、最大400塩基程度の配列を高効率にノックインする技術の開発に成功しており、今回その技術を発展させることにより、より長い配列(最大1,500塩基程度)の挿入に応用することで、ノックインマウスやコンディショナルノックアウトマウスとして使用されるfloxマウス*5の作製に成功しました。
また、その際に従来のCas9 mRNAとsgRNAを用いる方法ではなく、Cas9タンパク質とcrRNA/tracrRNAのCRISPRリボ核タンパク質(ctRNPと命名)をツールとして用いる方法を適用することにより、さらなる効率の改善が見られました。本研究グループでは、この新たな手法を「Efficient additions with ssDNA inserts-CRISPR(Easi-CRISPR)法」と命名しました。
実際に、この手法によって7種類の遺伝子のfloxマウス、6種類の遺伝子座位へのノックインマウスの作製を試み、全ての実験で目的のマウスを作製することに成功しています。
その遺伝子挿入効率は生まれてきたマウスのうち、floxマウスで8.5~100%、ノックインマウスで25~67%と極めて高いことが確認されています。
また、本手法では、平均40~60個の受精卵に顕微注入すれば目的のマウスが得られるのに対し、従来の手法では200~300個は受精卵が必要であり、それでも目的のマウスが得られないことも多々みられたことを考えると、作製効率の大幅な改善に成功したといえます。
さらに、本研究は、日本と米国の国際共同研究であり、マウス作製は三か所(東海大学、ネブラスカ大学、東京医科歯科大学)の独立した施設で行われました。いずれの施設でも本手法を用いて簡便に目的のマウスが作製できたことから、非常に再現性の高い手法であるといえます。
またEasi-CRISPR法は、マウス以外の生物種での遺伝子改変や、将来的な遺伝子治療への応用なども期待されるものです。
【用語解説】
◇CRISPRゲノム編集法*1
細菌などが持つ適応免疫に由来するシステム(CRISPR: clustered regularly interspaced short palindromic repeats)を応用した遺伝子改変技術です。CRISPRの構成要素であるCas9 DNA切断酵素と、標的領域に相補的な配列を含む短いRNA(sgRNAなど)を用いて、ゲノム上の任意の領域に二本鎖DNA切断を特異的に導入することができます。切断されたDNAは、細胞内に備わっているDNA修復系を用いて修復されようとしますが、その際に標的領域を自在に改変(削除、置換、挿入など)することが可能です。
◇一本鎖DNA*2
通常DNAは相補的な配列を持つ二本の鎖が対合した二重らせん構造を形成していますが、片方の鎖のみであり二重らせん構造を形成していないDNAを一本鎖DNAと呼びます。
◇二本鎖DNA*3
相補的な配列を持つ二本の鎖が対合した二重らせん構造を形成している状態のDNAです。
◇相同領域*4
目的DNA配列を含むドナーDNA(導入するDNA)の両端に付加する配列で、ゲノム上の標的領域の塩基配列と同じ塩基配列を持つDNA領域です。標的領域のDNAがCRISPRによって切断された後に、ドナーDNAの両端のDNA配列と標的領域のDNA配列との相同性を利用して、目的DNA配列が標的領域に挿入されます。
◇floxマウス*5
ノックアウト(遺伝子破壊)する標的遺伝子領域をCre組換え酵素の認識配列であるloxP配列で挟んだ遺伝子座を持つマウスです。組織特異的にCre酵素を発現するマウスと掛け合わせて、組織・時期特異的に目的遺伝子が破壊されたコンディショナルノックアウトマウスとして利用されます。
■本件に関するお問い合わせ
学校法人東海大学 経営企画室広報課
TEL:03-3467-2211(代表)
FAX:03-3485-4939
mail:pr@tokai.ac.jp
なお、本研究成果は5月17日(水)午前1時(イギリス時間)のオンラインジャーナル「Genome Biology」(DOI: 10.1186/s13059-017-1220-4)に掲載されました。
■研究の背景について
遺伝子改変マウスは、個体レベルでの遺伝子機能解析実験や疾患モデル動物として使用される極めて重要なツールの一つです。中でも最も使用頻度の高いマウスは、レポーター遺伝子や組み換え酵素遺伝子などを挿入した「ノックインマウス」、組織・時期特異的に遺伝子を破壊した「コンディショナルノックアウトマウス」であり、作製される全てのマウスの大半をこの二つのマウスが占めています。
これらのマウスを作製するには、標的とする遺伝子座位に対して目的のDNA配列を挿入(ノックイン)する必要があり、最近までES細胞を介した「遺伝子ターゲティング法」によるノックインマウスの作製が主流となっていましたが、この手法では時間とコスト、そして労力を要することが大きな課題となっていました。
こうした課題を受け、近年開発された「CRISPRゲノム編集法」は、受精卵に溶液を注入するだけで、ES細胞を介することなく簡便に目的とする遺伝子改変マウスを作製することが可能な画期的な手法です。しかし、この手法では単純なノックアウトマウスは高効率で作製できる一方で、ノックインマウスの作製効率は優れた技術を有する施設でも高々10%程度、コンディショナルノックアウトマウスでは数%程度と低く、その効率向上が課題となっていました。
「CRISPRゲノム編集法」では、遺伝子をノックインする際に用いる導入DNAに一本鎖DNA*2を用いると効率が高くなりますが、入手可能な一本鎖DNAの長さは200塩基までであり、それ以上の長さの配列を挿入するためには二本鎖DNA*3を用いる必要がありました。しかし、二本鎖DNAを用いた場合のノックイン効率は低く、また比較的長い相同領域*4を付加する必要があることから、ベクターの作製にも労力を要するという課題があります。
ノックイン効率の向上という課題の解消を目指して、いくつかの方法が世界中の様々な研究者によって開発されてきましたが、実用化に至る水準に達しておらず、この課題は遺伝子改変マウス作製に関わる研究分野全体の大きな問題となっており、新たな手法の確立が望まれていました。
■今回の研究成果について
今回、本研究グループでは、長鎖一本鎖DNAとCRISPRリボ核タンパク質を用いることで、受精卵への顕微注入を介したノックイン効率の大幅な向上に成功しました。長鎖一本鎖DNAは、挿入したいDNA配列を有するドナーDNAであり、目的DNA配列の両端に短い相同領域(50~100塩基程度)が付加されています。
本研究グループでは、長い一本鎖DNAを合成する方法を開発し、最大400塩基程度の配列を高効率にノックインする技術の開発に成功しており、今回その技術を発展させることにより、より長い配列(最大1,500塩基程度)の挿入に応用することで、ノックインマウスやコンディショナルノックアウトマウスとして使用されるfloxマウス*5の作製に成功しました。
また、その際に従来のCas9 mRNAとsgRNAを用いる方法ではなく、Cas9タンパク質とcrRNA/tracrRNAのCRISPRリボ核タンパク質(ctRNPと命名)をツールとして用いる方法を適用することにより、さらなる効率の改善が見られました。本研究グループでは、この新たな手法を「Efficient additions with ssDNA inserts-CRISPR(Easi-CRISPR)法」と命名しました。
実際に、この手法によって7種類の遺伝子のfloxマウス、6種類の遺伝子座位へのノックインマウスの作製を試み、全ての実験で目的のマウスを作製することに成功しています。
その遺伝子挿入効率は生まれてきたマウスのうち、floxマウスで8.5~100%、ノックインマウスで25~67%と極めて高いことが確認されています。
また、本手法では、平均40~60個の受精卵に顕微注入すれば目的のマウスが得られるのに対し、従来の手法では200~300個は受精卵が必要であり、それでも目的のマウスが得られないことも多々みられたことを考えると、作製効率の大幅な改善に成功したといえます。
さらに、本研究は、日本と米国の国際共同研究であり、マウス作製は三か所(東海大学、ネブラスカ大学、東京医科歯科大学)の独立した施設で行われました。いずれの施設でも本手法を用いて簡便に目的のマウスが作製できたことから、非常に再現性の高い手法であるといえます。
またEasi-CRISPR法は、マウス以外の生物種での遺伝子改変や、将来的な遺伝子治療への応用なども期待されるものです。
【用語解説】
◇CRISPRゲノム編集法*1
細菌などが持つ適応免疫に由来するシステム(CRISPR: clustered regularly interspaced short palindromic repeats)を応用した遺伝子改変技術です。CRISPRの構成要素であるCas9 DNA切断酵素と、標的領域に相補的な配列を含む短いRNA(sgRNAなど)を用いて、ゲノム上の任意の領域に二本鎖DNA切断を特異的に導入することができます。切断されたDNAは、細胞内に備わっているDNA修復系を用いて修復されようとしますが、その際に標的領域を自在に改変(削除、置換、挿入など)することが可能です。
◇一本鎖DNA*2
通常DNAは相補的な配列を持つ二本の鎖が対合した二重らせん構造を形成していますが、片方の鎖のみであり二重らせん構造を形成していないDNAを一本鎖DNAと呼びます。
◇二本鎖DNA*3
相補的な配列を持つ二本の鎖が対合した二重らせん構造を形成している状態のDNAです。
◇相同領域*4
目的DNA配列を含むドナーDNA(導入するDNA)の両端に付加する配列で、ゲノム上の標的領域の塩基配列と同じ塩基配列を持つDNA領域です。標的領域のDNAがCRISPRによって切断された後に、ドナーDNAの両端のDNA配列と標的領域のDNA配列との相同性を利用して、目的DNA配列が標的領域に挿入されます。
◇floxマウス*5
ノックアウト(遺伝子破壊)する標的遺伝子領域をCre組換え酵素の認識配列であるloxP配列で挟んだ遺伝子座を持つマウスです。組織特異的にCre酵素を発現するマウスと掛け合わせて、組織・時期特異的に目的遺伝子が破壊されたコンディショナルノックアウトマウスとして利用されます。
■本件に関するお問い合わせ
学校法人東海大学 経営企画室広報課
TEL:03-3467-2211(代表)
FAX:03-3485-4939
mail:pr@tokai.ac.jp

